Isochores¶
In [1]:
%config InlineBackend.close_figures=False
%matplotlib inline
from matplotlib import interactive
interactive(False)
In [2]:
from sur import *
setup_database()
In [3]:
m = Mixture()
m.add_many('methane propane n-pentane n-decane n-hexadecane',
'0.822 0.088 0.050 0.020 0.020')
s = EosSetup.objects.create(eos='RKPR', kij_mode=EosSetup.T_DEP, lij_mode='constants')
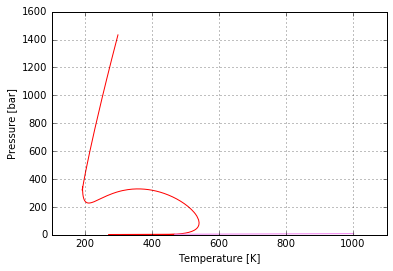
env = m.get_envelope(setup=s)
In [4]:
fig = env.plot()
fig
Out[4]:
In [15]:
help(m.get_isochore)
Help on method get_isochore in module sur.models:
get_isochore(self, setup, v, ts, ps, t_sup, t_step=5.0, t_inf=270.0) method of sur.models.Mixture instance
Get the isochore (isoV) for this mixture, calculated using
the setup EOS with its selected interaction parameters
mode.
In [5]:
isochore = m.get_isochore(setup=s, v=10., ts=467.01, ps=3.86, t_sup=465.0, t_step=5.0, t_inf=270.0)
You can get the raw input and output
In [6]:
print(isochore.input_txt)
5 NC
5 nplus
isoV Spec v(k), ts and ps
0.822 0.088 0.05 0.02 0.02 z1,z2...zNC
10.0 467.01 3.86 v(L/mol)+Ts+Ps
465.0 5.0 270.0 T(K)+dT+Tinf
3 NMODEL (1:SRK / 2:PR / 3:RKPR)
0 1 ncomb, nTdep
METHANE(1)
190.564 45.99 0.0115478 0.116530154855 1.16 tc, pc, ohm, vc, zrat
2.30376807604 0.0304337956072 0.5 1.54083758839 ac, b, delta1, k
PROPANE(2)
369.83 42.48 0.152291 0.233012170918 1.16 tc, pc, ohm, vc, zrat
9.80216972295 0.0598748909487 1.663687 1.9574557212 ac, b, delta1, k
0.013831 k0
190.564 tstar
-0.047134 lij
n-PENTANE(3)
469.7 33.7 0.251506 0.366506659349 1.16 tc, pc, ohm, vc, zrat
20.2236971345 0.0936309815449 1.957315 2.28798764446 ac, b, delta1, k
0.030221 0.0 k0
190.564 369.83 tstar
-0.084739 -0.010835 lij
n-DECANE(4)
617.7 21.1 0.492328 0.756468369911 1.16 tc, pc, ohm, vc, zrat
56.6610724692 0.192140038284 2.239538 3.11337933794 ac, b, delta1, k
0.068534 0.0 0.0 k0
190.564 369.83 469.7 tstar
-0.145519 -0.032481 -0.02353 lij
n-HEXADECANE(5)
723.0 14.0 0.717404 1.34252502769 1.16 tc, pc, ohm, vc, zrat
116.426356444 0.341676477137 2.14291 3.90352446586 ac, b, delta1, k
0.095928 0.0 0.0 0.0 k0
190.564 369.83 469.7 617.7 tstar
-0.121142 -0.023269 -0.012501 0.049878 lij
In [7]:
print(isochore.output_txt)
The plus fraction from component 5 is 2.000000000000000E-002
T(K) rho(mol/L) P(bar) v(L/mol) beta betav xplus yplus
iter
467.01 0.1000E+00 0.3860E+01 0.1000E+02 0.1000E+01 0.1000E+01
465.00 0.1000E+00 0.3845E+01 0.1000E+02 0.9984E+00 0.9999E+00 0.898332 0.018611 6
460.00 0.1000E+00 0.3792E+01 0.1000E+02 0.9950E+00 0.9998E+00 0.890882 0.015582 7
455.00 0.1000E+00 0.3740E+01 0.1000E+02 0.9919E+00 0.9997E+00 0.882580 0.012958 7
450.00 0.1000E+00 0.3689E+01 0.1000E+02 0.9892E+00 0.9996E+00 0.873347 0.010699 7
445.00 0.1000E+00 0.3640E+01 0.1000E+02 0.9869E+00 0.9995E+00 0.863099 0.008769 7
440.00 0.1000E+00 0.3591E+01 0.1000E+02 0.9848E+00 0.9994E+00 0.851757 0.007130 7
435.00 0.1000E+00 0.3544E+01 0.1000E+02 0.9829E+00 0.9994E+00 0.839247 0.005750 8
430.00 0.1000E+00 0.3498E+01 0.1000E+02 0.9812E+00 0.9993E+00 0.825509 0.004596 8
425.00 0.1000E+00 0.3451E+01 0.1000E+02 0.9797E+00 0.9993E+00 0.810500 0.003639 8
420.00 0.1000E+00 0.3406E+01 0.1000E+02 0.9783E+00 0.9992E+00 0.794204 0.002854 8
415.00 0.1000E+00 0.3361E+01 0.1000E+02 0.9770E+00 0.9992E+00 0.776641 0.002215 9
410.00 0.1000E+00 0.3316E+01 0.1000E+02 0.9758E+00 0.9992E+00 0.757877 0.001701 9
405.00 0.1000E+00 0.3271E+01 0.1000E+02 0.9746E+00 0.9991E+00 0.738030 0.001291 9
400.00 0.1000E+00 0.3227E+01 0.1000E+02 0.9734E+00 0.9991E+00 0.717280 0.000969 9
395.00 0.1000E+00 0.3182E+01 0.1000E+02 0.9723E+00 0.9991E+00 0.695871 0.000719 9
390.00 0.1000E+00 0.3138E+01 0.1000E+02 0.9711E+00 0.9991E+00 0.674107 0.000527 9
385.00 0.1000E+00 0.3094E+01 0.1000E+02 0.9699E+00 0.9990E+00 0.652340 0.000382 9
380.00 0.1000E+00 0.3050E+01 0.1000E+02 0.9687E+00 0.9990E+00 0.630950 0.000273 8
375.00 0.1000E+00 0.3006E+01 0.1000E+02 0.9675E+00 0.9990E+00 0.610313 0.000193 8
370.00 0.1000E+00 0.2962E+01 0.1000E+02 0.9664E+00 0.9990E+00 0.590768 0.000136 8
365.00 0.1000E+00 0.2918E+01 0.1000E+02 0.9652E+00 0.9990E+00 0.572588 0.000094 7
360.00 0.1000E+00 0.2874E+01 0.1000E+02 0.9641E+00 0.9989E+00 0.555955 0.000065 7
355.00 0.1000E+00 0.2831E+01 0.1000E+02 0.9631E+00 0.9989E+00 0.540952 0.000044 7
350.00 0.1000E+00 0.2788E+01 0.1000E+02 0.9621E+00 0.9989E+00 0.527568 0.000030 6
345.00 0.1000E+00 0.2745E+01 0.1000E+02 0.9613E+00 0.9989E+00 0.515708 0.000020 6
340.00 0.1000E+00 0.2703E+01 0.1000E+02 0.9604E+00 0.9989E+00 0.505214 0.000013 6
335.00 0.1000E+00 0.2660E+01 0.1000E+02 0.9597E+00 0.9989E+00 0.495883 0.000008 6
330.00 0.1000E+00 0.2618E+01 0.1000E+02 0.9590E+00 0.9989E+00 0.487479 0.000005 6
325.00 0.1000E+00 0.2576E+01 0.1000E+02 0.9583E+00 0.9988E+00 0.479748 0.000003 6
320.00 0.1000E+00 0.2534E+01 0.1000E+02 0.9577E+00 0.9988E+00 0.472422 0.000002 6
315.00 0.1000E+00 0.2493E+01 0.1000E+02 0.9570E+00 0.9988E+00 0.465218 0.000001 7
310.00 0.1000E+00 0.2451E+01 0.1000E+02 0.9563E+00 0.9988E+00 0.457840 0.000001 7
305.00 0.1000E+00 0.2409E+01 0.1000E+02 0.9556E+00 0.9988E+00 0.449965 0.000000 7
300.00 0.1000E+00 0.2366E+01 0.1000E+02 0.9547E+00 0.9988E+00 0.441244 0.000000 7
295.00 0.1000E+00 0.2324E+01 0.1000E+02 0.9536E+00 0.9988E+00 0.431283 0.000000 8
290.00 0.1000E+00 0.2281E+01 0.1000E+02 0.9523E+00 0.9988E+00 0.419647 0.000000 8
285.00 0.1000E+00 0.2237E+01 0.1000E+02 0.9507E+00 0.9988E+00 0.405877 0.000000 8
280.00 0.1000E+00 0.2193E+01 0.1000E+02 0.9487E+00 0.9987E+00 0.389552 0.000000 8
275.00 0.1000E+00 0.2147E+01 0.1000E+02 0.9460E+00 0.9987E+00 0.370439 0.000000 9
270.00 0.1000E+00 0.2100E+01 0.1000E+02 0.9427E+00 0.9987E+00 0.348747 0.000000 9
Monophasic Region:
T(K) rho(mol/L) P(bar) v(L/mol)
467.01 0.1000E+00 0.3860E+01 0.1000E+02
472.01 0.1000E+00 0.3910E+01 0.1000E+02
477.01 0.1000E+00 0.3952E+01 0.1000E+02
482.01 0.1000E+00 0.3994E+01 0.1000E+02
487.01 0.1000E+00 0.4036E+01 0.1000E+02
492.01 0.1000E+00 0.4078E+01 0.1000E+02
497.01 0.1000E+00 0.4120E+01 0.1000E+02
502.01 0.1000E+00 0.4162E+01 0.1000E+02
507.01 0.1000E+00 0.4204E+01 0.1000E+02
512.01 0.1000E+00 0.4246E+01 0.1000E+02
517.01 0.1000E+00 0.4288E+01 0.1000E+02
522.01 0.1000E+00 0.4330E+01 0.1000E+02
527.01 0.1000E+00 0.4372E+01 0.1000E+02
532.01 0.1000E+00 0.4414E+01 0.1000E+02
537.01 0.1000E+00 0.4456E+01 0.1000E+02
542.01 0.1000E+00 0.4498E+01 0.1000E+02
547.01 0.1000E+00 0.4540E+01 0.1000E+02
552.01 0.1000E+00 0.4582E+01 0.1000E+02
557.01 0.1000E+00 0.4624E+01 0.1000E+02
562.01 0.1000E+00 0.4666E+01 0.1000E+02
567.01 0.1000E+00 0.4708E+01 0.1000E+02
572.01 0.1000E+00 0.4750E+01 0.1000E+02
577.01 0.1000E+00 0.4792E+01 0.1000E+02
582.01 0.1000E+00 0.4834E+01 0.1000E+02
587.01 0.1000E+00 0.4876E+01 0.1000E+02
592.01 0.1000E+00 0.4918E+01 0.1000E+02
597.01 0.1000E+00 0.4960E+01 0.1000E+02
602.01 0.1000E+00 0.5002E+01 0.1000E+02
607.01 0.1000E+00 0.5044E+01 0.1000E+02
612.01 0.1000E+00 0.5086E+01 0.1000E+02
617.01 0.1000E+00 0.5128E+01 0.1000E+02
622.01 0.1000E+00 0.5170E+01 0.1000E+02
627.01 0.1000E+00 0.5212E+01 0.1000E+02
632.01 0.1000E+00 0.5254E+01 0.1000E+02
637.01 0.1000E+00 0.5296E+01 0.1000E+02
642.01 0.1000E+00 0.5338E+01 0.1000E+02
647.01 0.1000E+00 0.5380E+01 0.1000E+02
652.01 0.1000E+00 0.5422E+01 0.1000E+02
657.01 0.1000E+00 0.5464E+01 0.1000E+02
662.01 0.1000E+00 0.5506E+01 0.1000E+02
667.01 0.1000E+00 0.5548E+01 0.1000E+02
672.01 0.1000E+00 0.5590E+01 0.1000E+02
677.01 0.1000E+00 0.5632E+01 0.1000E+02
682.01 0.1000E+00 0.5674E+01 0.1000E+02
687.01 0.1000E+00 0.5716E+01 0.1000E+02
692.01 0.1000E+00 0.5758E+01 0.1000E+02
697.01 0.1000E+00 0.5800E+01 0.1000E+02
702.01 0.1000E+00 0.5842E+01 0.1000E+02
707.01 0.1000E+00 0.5884E+01 0.1000E+02
712.01 0.1000E+00 0.5926E+01 0.1000E+02
717.01 0.1000E+00 0.5967E+01 0.1000E+02
722.01 0.1000E+00 0.6009E+01 0.1000E+02
727.01 0.1000E+00 0.6051E+01 0.1000E+02
732.01 0.1000E+00 0.6093E+01 0.1000E+02
737.01 0.1000E+00 0.6135E+01 0.1000E+02
742.01 0.1000E+00 0.6177E+01 0.1000E+02
747.01 0.1000E+00 0.6219E+01 0.1000E+02
752.01 0.1000E+00 0.6261E+01 0.1000E+02
757.01 0.1000E+00 0.6303E+01 0.1000E+02
762.01 0.1000E+00 0.6345E+01 0.1000E+02
767.01 0.1000E+00 0.6387E+01 0.1000E+02
772.01 0.1000E+00 0.6429E+01 0.1000E+02
777.01 0.1000E+00 0.6471E+01 0.1000E+02
782.01 0.1000E+00 0.6513E+01 0.1000E+02
787.01 0.1000E+00 0.6554E+01 0.1000E+02
792.01 0.1000E+00 0.6596E+01 0.1000E+02
797.01 0.1000E+00 0.6638E+01 0.1000E+02
802.01 0.1000E+00 0.6680E+01 0.1000E+02
807.01 0.1000E+00 0.6722E+01 0.1000E+02
812.01 0.1000E+00 0.6764E+01 0.1000E+02
817.01 0.1000E+00 0.6806E+01 0.1000E+02
822.01 0.1000E+00 0.6848E+01 0.1000E+02
827.01 0.1000E+00 0.6890E+01 0.1000E+02
832.01 0.1000E+00 0.6932E+01 0.1000E+02
837.01 0.1000E+00 0.6974E+01 0.1000E+02
842.01 0.1000E+00 0.7015E+01 0.1000E+02
847.01 0.1000E+00 0.7057E+01 0.1000E+02
852.01 0.1000E+00 0.7099E+01 0.1000E+02
857.01 0.1000E+00 0.7141E+01 0.1000E+02
862.01 0.1000E+00 0.7183E+01 0.1000E+02
867.01 0.1000E+00 0.7225E+01 0.1000E+02
872.01 0.1000E+00 0.7267E+01 0.1000E+02
877.01 0.1000E+00 0.7309E+01 0.1000E+02
882.01 0.1000E+00 0.7351E+01 0.1000E+02
887.01 0.1000E+00 0.7392E+01 0.1000E+02
892.01 0.1000E+00 0.7434E+01 0.1000E+02
897.01 0.1000E+00 0.7476E+01 0.1000E+02
902.01 0.1000E+00 0.7518E+01 0.1000E+02
907.01 0.1000E+00 0.7560E+01 0.1000E+02
912.01 0.1000E+00 0.7602E+01 0.1000E+02
917.01 0.1000E+00 0.7644E+01 0.1000E+02
922.01 0.1000E+00 0.7686E+01 0.1000E+02
927.01 0.1000E+00 0.7728E+01 0.1000E+02
932.01 0.1000E+00 0.7769E+01 0.1000E+02
937.01 0.1000E+00 0.7811E+01 0.1000E+02
942.01 0.1000E+00 0.7853E+01 0.1000E+02
947.01 0.1000E+00 0.7895E+01 0.1000E+02
952.01 0.1000E+00 0.7937E+01 0.1000E+02
957.01 0.1000E+00 0.7979E+01 0.1000E+02
962.01 0.1000E+00 0.8021E+01 0.1000E+02
967.01 0.1000E+00 0.8063E+01 0.1000E+02
972.01 0.1000E+00 0.8104E+01 0.1000E+02
977.01 0.1000E+00 0.8146E+01 0.1000E+02
982.01 0.1000E+00 0.8188E+01 0.1000E+02
987.01 0.1000E+00 0.8230E+01 0.1000E+02
992.01 0.1000E+00 0.8272E+01 0.1000E+02
997.01 0.1000E+00 0.8314E+01 0.1000E+02
1002.01 0.1000E+00 0.8356E+01 0.1000E+02
los vectores t y p son recortados en dos partes, como se generan
In [8]:
isochore.t
Out[8]:
array([ 467.01, 465. , 460. , 455. , 450. , 445. , 440. ,
435. , 430. , 425. , 420. , 415. , 410. , 405. ,
400. , 395. , 390. , 385. , 380. , 375. , 370. ,
365. , 360. , 355. , 350. , 345. , 340. , 335. ,
330. , 325. , 320. , 315. , 310. , 305. , 300. ,
295. , 290. , 285. , 280. , 275. , 270. ])
In [9]:
isochore.p
Out[9]:
array([ 3.86 , 3.845, 3.792, 3.74 , 3.689, 3.64 , 3.591, 3.544,
3.498, 3.451, 3.406, 3.361, 3.316, 3.271, 3.227, 3.182,
3.138, 3.094, 3.05 , 3.006, 2.962, 2.918, 2.874, 2.831,
2.788, 2.745, 2.703, 2.66 , 2.618, 2.576, 2.534, 2.493,
2.451, 2.409, 2.366, 2.324, 2.281, 2.237, 2.193, 2.147, 2.1 ])
In [10]:
isochore.t_monophasic
Out[10]:
array([ 467.01, 472.01, 477.01, 482.01, 487.01, 492.01,
497.01, 502.01, 507.01, 512.01, 517.01, 522.01,
527.01, 532.01, 537.01, 542.01, 547.01, 552.01,
557.01, 562.01, 567.01, 572.01, 577.01, 582.01,
587.01, 592.01, 597.01, 602.01, 607.01, 612.01,
617.01, 622.01, 627.01, 632.01, 637.01, 642.01,
647.01, 652.01, 657.01, 662.01, 667.01, 672.01,
677.01, 682.01, 687.01, 692.01, 697.01, 702.01,
707.01, 712.01, 717.01, 722.01, 727.01, 732.01,
737.01, 742.01, 747.01, 752.01, 757.01, 762.01,
767.01, 772.01, 777.01, 782.01, 787.01, 792.01,
797.01, 802.01, 807.01, 812.01, 817.01, 822.01,
827.01, 832.01, 837.01, 842.01, 847.01, 852.01,
857.01, 862.01, 867.01, 872.01, 877.01, 882.01,
887.01, 892.01, 897.01, 902.01, 907.01, 912.01,
917.01, 922.01, 927.01, 932.01, 937.01, 942.01,
947.01, 952.01, 957.01, 962.01, 967.01, 972.01,
977.01, 982.01, 987.01, 992.01, 997.01, 1002.01])
In [11]:
isochore.p_monophasic
Out[11]:
array([ 3.86 , 3.91 , 3.952, 3.994, 4.036, 4.078, 4.12 , 4.162,
4.204, 4.246, 4.288, 4.33 , 4.372, 4.414, 4.456, 4.498,
4.54 , 4.582, 4.624, 4.666, 4.708, 4.75 , 4.792, 4.834,
4.876, 4.918, 4.96 , 5.002, 5.044, 5.086, 5.128, 5.17 ,
5.212, 5.254, 5.296, 5.338, 5.38 , 5.422, 5.464, 5.506,
5.548, 5.59 , 5.632, 5.674, 5.716, 5.758, 5.8 , 5.842,
5.884, 5.926, 5.967, 6.009, 6.051, 6.093, 6.135, 6.177,
6.219, 6.261, 6.303, 6.345, 6.387, 6.429, 6.471, 6.513,
6.554, 6.596, 6.638, 6.68 , 6.722, 6.764, 6.806, 6.848,
6.89 , 6.932, 6.974, 7.015, 7.057, 7.099, 7.141, 7.183,
7.225, 7.267, 7.309, 7.351, 7.392, 7.434, 7.476, 7.518,
7.56 , 7.602, 7.644, 7.686, 7.728, 7.769, 7.811, 7.853,
7.895, 7.937, 7.979, 8.021, 8.063, 8.104, 8.146, 8.188,
8.23 , 8.272, 8.314, 8.356])
In [12]:
isochore.rho, isochore.rho_monophasic,
Out[12]:
(array([ 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1]),
array([ 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1]))
In [13]:
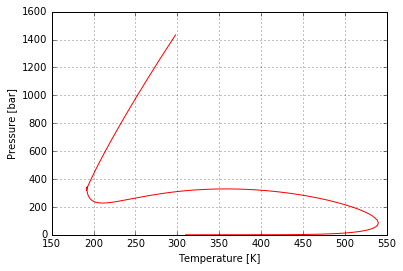
isochore.plot()
Out[13]:
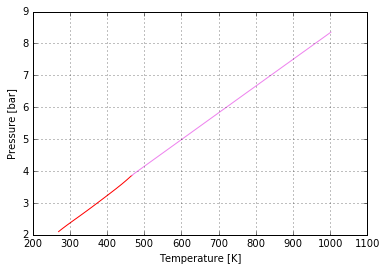
También se le puede pasar una figura ya generada, para que superponga el nuevo plot
In [14]:
isochore.plot(fig)
Out[14]: